Molt bons resultats del grup de recerca en Ciència Computacional i Acellera al D3R Grand Challenge 4
Molt bons resultats del grup de recerca en Ciència Computacional i Acellera al D3R Grand Challenge 4
Han quedat classificats en primer lloc en dos dels desafiaments del concurs D3R Grand Challenge 4.
L'estiu passat, Acellera i el Computational Science Lab van decidir participar en el D3R Grand Challenge 4, un concurs internacional que consisteix a completar diversos desafiaments considerats
d'interès farmacèutic. En general, aquestes tasques impliquen predir com un fàrmac s'uneix a una proteïna i/o com de fort és aquesta unió. Idealment, si es poguessin predir aquests valors de manera precisa, s'acceleraria el procés de descobriment de nous medicaments i també disminuirien els costos de desenvolupar nous fàrmacs. Per tant, un dels aspectes més interessants d'aquests desafiaments és que poden proporcionar una mesura aproximada de com des de lluny estem d'aquesta situació ideal.
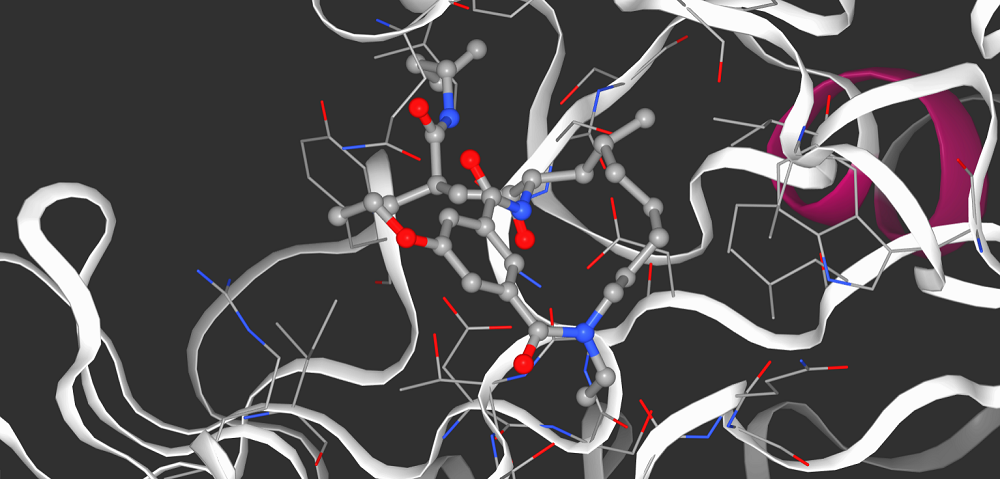
L'empresa Acellera i el grup de recerca van treballar junts en aquest projecte, utilitzant algunes eines que ja estaven disponibles a PlayMolecule, com KDeep o DeltaDelta, i desenvolupant una nova: SkeleDock. KDeep i DeltaDelta poden fer prediccions precises de la força d'unió que esmentem, però necessiten “veure” la interacció entre el fàrmac candidat i la proteïna. A quina part de la proteïna s'uneix el fàrmac? En quina orientació es col·loca la molècula dins de la butxaca proteica? Aquestes són les preguntes que SkeleDock intenta respondre, utilitzant el coneixement estructural previ disponible en bases de dades com el Protein Data Bank.
Després d'afrontar diverses dificultats en el desenvolupament de SkeleDock, vam poder proporcionar, amb cert nivell de precisió, respostes a aquestes preguntes, generant imatges en 3D que capturen el lligand dins la butxaca de la proteïna. Després, vam proporcionar aquestes imatges en 3D a KDeep, de manera que pogués fer prediccions sobre la força de la unió. Finalment, vam presentar aquestes prediccions.
La setmana passada, els organitzadors del D3R van anunciar els resultats per als diferents desafiaments del concurs. Ens complau informar que hem quedat classificats en primer lloc en dos d'ells (predicció de l'energia lliure de diversos ligands a la proteïna BACE, i un rànquing d'un set més gran de ligands per a aquesta mateixa proteïna, tots dos en l'etapa 2), així com en la sisena posició (segons la mètrica utilitzada) en el desafiament de predicció de manera d'unió lligant-proteïna entre més de 80 participants. No obstant això, en altres tasques en les quals vam participar no vam obtenir tan bons resultats; concretament, en aquelles que implicaven la proteïna Catepsina S. En aquest cas, les molècules d'aigua semblen jugar un paper molt important que, de moment, ni SkeleDock ni KDeep tenen la capacitat de tenir en compte. Treballarem en aquests aspectes per a millorar les nostres eines i, amb sort, podrem obtenir millors resultats la pròxima vegada.
SkeleDock aviat estarà disponible a PlayMolecule per a tothom i es publicarà un article amb més detalls dels protocols que utilitzem.
Podeu trobar més informació al blog d’Acellera.
