Muy buenos resultados del grupo de investigación en Ciencia Computacional y Acellera en el D3R Grand Challenge 4
Muy buenos resultados del grupo de investigación en Ciencia Computacional y Acellera en el D3R Grand Challenge 4
Han quedado clasificados en primer lugar en dos de los desafíos del concurso D3R Grand Challenge 4.
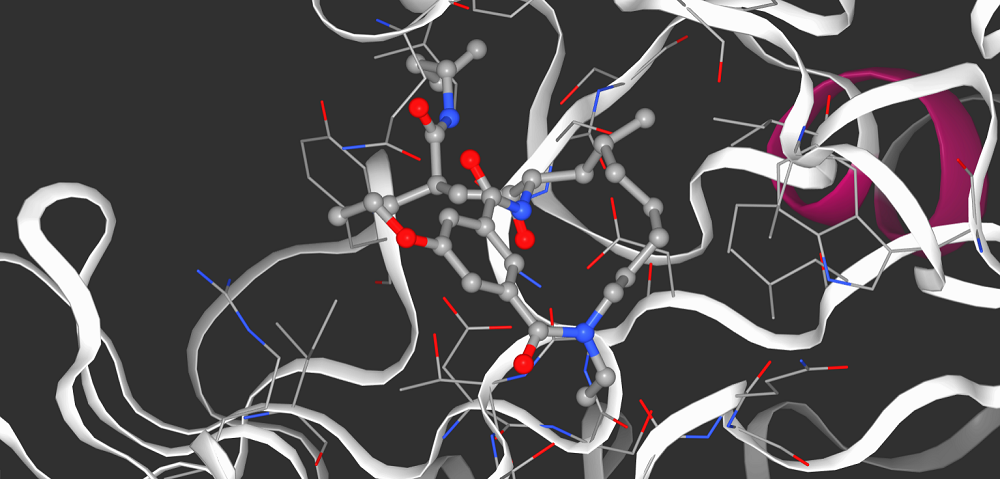
El verano pasado, Acellera y el Computational Science Lab decidieron participar en el D3R Grand Challenge 4, un concurso internacional que consiste en completar varios desafíos considerados de interés farmacéutico. Por lo general, estas tareas implican predecir cómo un fármaco se une a una proteína y/o cómo de fuerte es esta unión. Idealmente, si se pudieran predecir estos valores de manera precisa, se aceleraría el proceso de descubrimiento de nuevos medicamentos y también disminuirían los costes de desarrollar nuevos fármacos. Por lo tanto, uno de los aspectos más interesantes de estos desafíos es que pueden proporcionar una medida aproximada de cómo de lejos estamos de esta situación ideal.
La empresa Acellera y el grupo de investigación trabajaron juntos en este proyecto, utilizando algunas herramientas que ya estaban disponibles en PlayMolecule, como KDeep o DeltaDelta, y desarrollando una nueva: SkeleDock. KDeep y DeltaDelta pueden hacer predicciones precisas de la fuerza de unión que mencionamos, pero necesitan ver la interacción entre el fármaco candidato y la proteína. ¿En qué parte de la proteína se une el fármaco? ¿En qué orientación se coloca la molécula dentro del bolsillo proteico? Estas son las preguntas que SkeleDock intenta responder, utilizando el conocimiento estructural previo disponible en bases de datos como el Protein Data Bank.
Después de afrontar varias dificultades en el desarrollo de SkeleDock, pudimos proporcionar, con cierto nivel de precisión, respuestas a estas preguntas, generando imágenes en 3D que capturan el ligando dentro del bolsillo de la proteína. Después, proporcionamos estas imágenes en 3D a KDeep, de modo que pudiera hacer predicciones sobre la fuerza de la unión. Finalmente, presentamos estas predicciones.
La semana pasada, los organizadores del D3R anunciaron los resultados para los diferentes desafíos del concurso. Nos complace informar que hemos quedado clasificados en primer lugar en dos de ellos (predicción de la energía libre de varios ligandos en la proteína BACE, y un ranking de un set más grande de ligandos para esta misma proteína, ambos en la etapa 2), así como en la sexta posición (según la métrica utilizada) en el desafío de predicción de modo de unión ligando-proteína entre más de 80 participantes. Sin embargo, en otras tareas en las que participamos no obtuvimos tan buenos resultados; concretamente, en aquellas que implicaban a la proteína Catepsina S. En este caso, las moléculas de agua parecen jugar un papel muy importante que, por el momento, ni SkeleDock ni KDeep tienen la capacidad de tener en cuenta. Trabajaremos en estos aspectos para mejorar nuestras herramientas y, con suerte, podremos obtener mejores resultados la próxima vez.
SkeleDock pronto estará disponible en PlayMolecule para todo el mundo y se publicará un artículo con más detalles de los protocolos que utilizamos.
Podéis encontrar más información en el blog de Acellera.
