Un método de segmentación de imagen médica logra el mejor rendimiento en el Challenge NeoBrainS12
Desarrollado por investigadores del Grupo de Investigación Simbiosys, los autores lo describen en una comunicación presentada en el 7th International Workshop Machine Learning in Medical Imaging, celebrado el 17 de octubre en Atenas (Grecia).
Los tejidos y las estructuras cerebrales se estudian a partir de imágenes médicas que luego se procesan e interpretan a través de métodos de segmentación, con el objetivo de apoyar el diagnóstico basado en imágenes. Hoy en día, las dos metodologías más utilizadas en la segmentación de estructuras se basan en el modelado de intensidades de los tejidos y en la fusión multi-atlas.
La informática tiene un papel fundamental en el desarrollo de nuevas técnicas de adquisición y análisis cuantitativo de imágenes médicas obtenidas por resonancia magnética intracraneal (RMI). Un análisis que, aunque expertos en neuroimagen pueden hacer manualmente, es lento. De ahí que el desarrollo de métodos automáticos para caracterizar de manera precisa las principales estructuras cerebrales sea decisivo para ayudar al diagnóstico. La metodología automática es rápida, objetiva y consigue métodos reproducibles para una gran cantidad de datos.
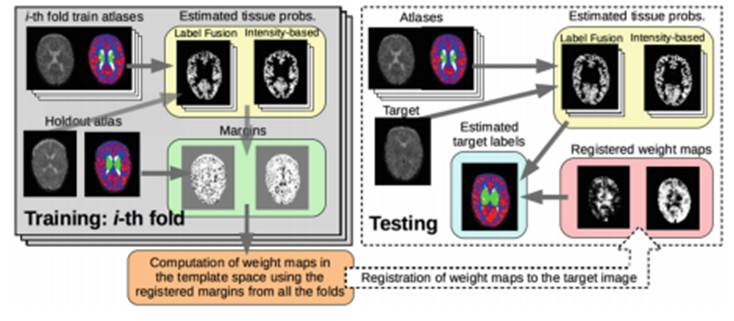
Investigadores Simbiosys (Simulation, Imaging and Modelling for Biomedical Systems),Grupo de Investigación del Departamento de Tecnologías de la Información y las Comunicaciones (DTIC) de la UPF, han elaborado un nuevo método de segmentación que combina características complementarias de los dos métodos habitualmente más empleados, citados anteriormente, y consiguiendo mejores resultados.
Gerard Sanroma, Oualid M. Benkarim, y Gemma Piella, investigadores del Grupo de Investigación Simbiosys, con Miguel Ángel González Ballester, jefe del grupo y profesor de investigación ICREA del DTIC, explican los detalles de este nuevo método en una comunicación que han presentado al 7th International Workshop Machine Learning in Medical Imaging, celebrado el 17 de octubre en Atenas ( Grecia).
Simbiosys participa en el Challenge NeoBrain12
El volumen de tejido cerebral neonatal se considera un buen indicador del rendimiento del desarrollo neurológico a largo plazo. La cuantificación del volumen de tejido cerebral se lleva a cabo mediante métodos de segmentación de imagen. Una aplicación práctica del algoritmo creado a Simbiosys radica en la participación del grupo en el Challenge NeoBrainS12. Como ha explicado Sanroma: "nuestro método de segmentación de imágenes de cerebros neonatales está superando el resto de métodos que se presentan al Challenge NeoBrainS12".
Coordinado desde los Países Bajos, el objetivo del NeoBrainS12 es comparar los rendimientos de los métodos de algoritmos automáticos de cuantificación aplicados a las imágenes de cerebros neonatales siguiendo unos determinados protocolos. En el NeoBrain12 diferentes grupos de trabajo proponen sus algoritmos que se comparan con el mismo patrón de referencia para identificar los de mejor rendimiento. Desde el 2013, el grupo Simbiosys está participando en este reto y los resultados indican que el método de segmentación de Sanroma et al., en muchas de las categorías estudiadas para el challenge, está alcanzando los primeros puestos, con mejoras de hasta un 10% en rendimiento.
Trabajo de referencia:
Gerard Sanroma, Oualid M. Benkarim, Gemma Piella, Miguel Ángel González Ballester (2016), “Building an Ensemble of Complementary Segmentation Methods by Exploiting Probabilistic Estimates”, pp. 27-25, en: L. Wang et al. (Eds): MLMI 2016, LNCS 10019, Springer International Publishing. DOI: 10.1007/978-3-319-47157-0_4
