El ADN de los primates revela aplicaciones para la salud humana
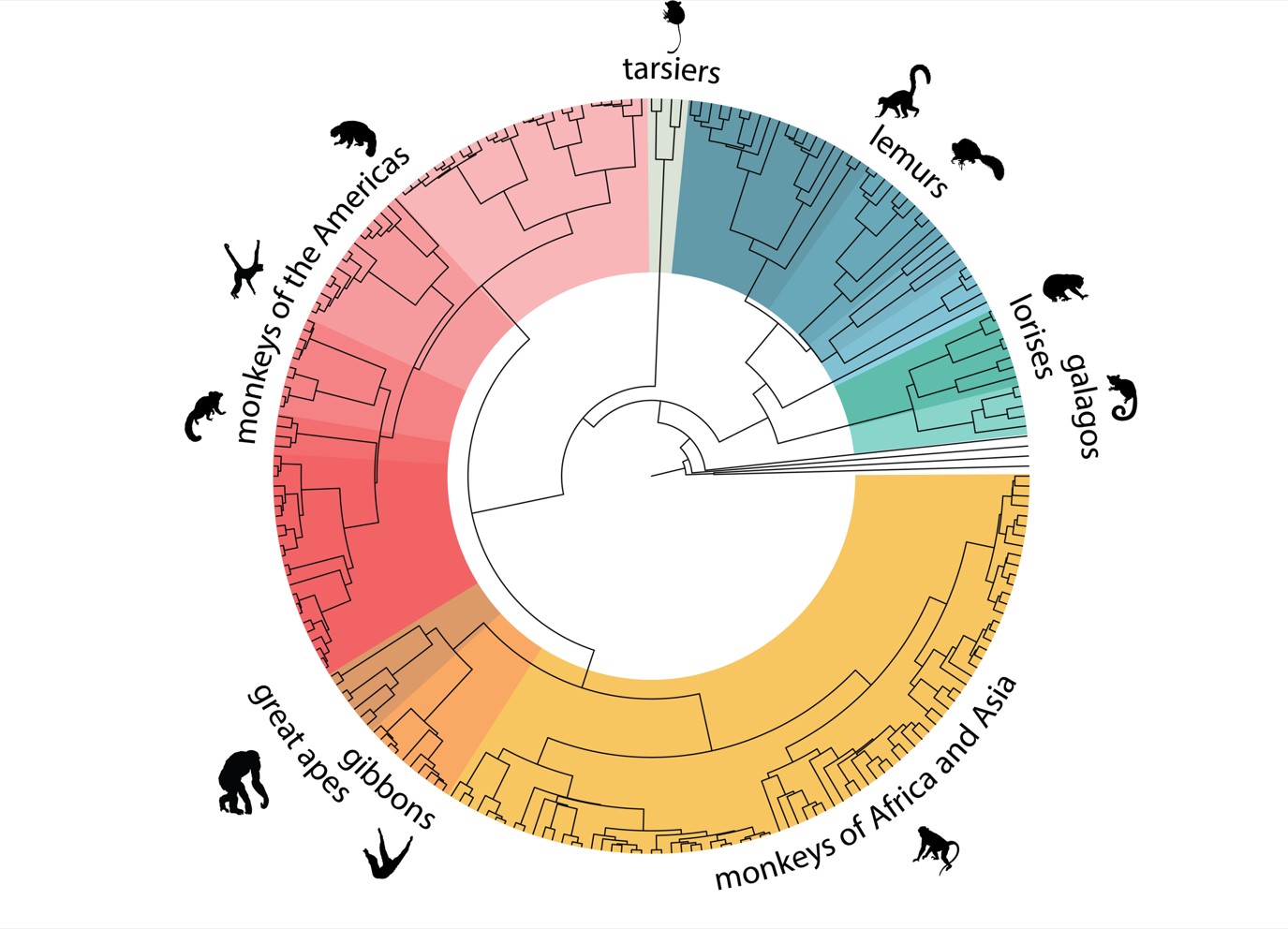
Los genomas de 233 especies de primates revelan características clave de la evolución de los humanos, las enfermedades humanas y la conservación de su biodiversidad. El Instituto de Biología Evolutiva (IBE, CSIC-UPF), la Universidad Pompeu Fabra, Illumina y la Facultad de Medicina de Baylor han liderado un número especial de Science, uno de los pocos producidos desde España.

Una nueva investigación liderada por Tomàs Marquès-Bonet, Investigador ICREA en el IBE (CSIC-UPF) y Catedrático de Genética del Departamento de Medicina y Ciencias de la Vida (MELIS) de la Universidad Pompeu Fabra (UPF); el Dr. Kyle Farh (Illumina) y el Dr. Jeffrey Rogers (Facultad de Medicina de Baylor), combina la secuenciación del genoma de más de 809 individuos de 233 especies de primates, cubriendo casi la mitad de todas las especies de primates existentes en la Tierra, incluyendo el estudio de restos fósiles y multiplicando por 4 el número de los genomas de primates disponibles hasta ahora.
El estudio proporciona nueva información sobre la diversidad genética y la filogenia de los primates, que es importante para comprender y conservar la diversidad de la especie más cercana a la nuestra. Del mismo modo, identifica una nueva forma de encontrar mutaciones genéticas que podrían ser responsables de muchas enfermedades humanas.
En uno de los pocos números especiales de la revista Science dirigidos desde España, se revelaron los genomas del 86% de los géneros y de todas las familias de primates. De ellos, 8 de cada 10 han sido secuenciados en el Centro Nacional de Análisis Genómico (CNAG) de Barcelona.
“Este proyecto ha sido uno de los más importantes en los que hemos trabajado en los últimos años en el CNAG. Hemos desarrollado una nueva metodología de secuenciación, analizando más de 600 genomas de primates. Sin duda, un estudio clave que nos ha permitido obtener una visión muy completa de la variación genética de los primates y del genoma humano”, explica Ivo Gut, director del CNAG.
Las mutaciones raras pueden aumentar el riesgo de enfermedad
Una de las limitaciones de la genética humana y clínica es la incapacidad actual para detectar, entre cientos de miles de mutaciones, las que causan enfermedades. Actualmente se desconocen las causas genéticas de muchas enfermedades comunes, como la diabetes y las cardiopatías, debido a la falta de información genética o al gran número de factores genéticos implicados. Se cree que algunas enfermedades se originan cuando un conjunto de variaciones genéticas o mutaciones con un efecto "leve" actúan conjuntamente para causar una enfermedad de origen poligénico, como la diabetes o el cáncer.
Al comparar el genoma de 809 individuos de 233 especies de primates no humanos con el humano, la investigación ha identificado 4,3 millones de mutaciones “missense” que afectan a la composición de los aminoácidos y pueden alterar la función de las proteínas. Estas mutaciones identificadas en primates son la base para realizar estudios comparativos con las variantes humanas e identificar aquellas variantes clave en muchas enfermedades humanas.
“El 6% de estas mutaciones son abundantes en primates y, por tanto, se consideran "potencialmente benignas" en enfermedades humanas, dado que se tolera su presencia en estos animales.”, afirma Kyle Farh, vicepresidente de Inteligencia Artificial de Illumina y coautor principal de las publicaciones.
La identificación de mutaciones causantes de enfermedades se ha logrado gracias al algoritmo de aprendizaje profundo PrimateAI-3D. Un algoritmo de inteligencia artificial (IA) desarrollado por Illumina, la empresa líder mundial en secuenciación de ADN, que es una especie de ChatGPT para genética que utiliza la secuencia del genoma en lugar del lenguaje humano.
Nuevos conocimientos sobre la evolución de los primates y la singularidad humana
La publicación de este proyecto contiene el catálogo más completo de información genómica de primates elaborado hasta la fecha, que abarca casi la mitad de todas las especies de primates existentes en la Tierra. Contiene información de primates de Asia, América, África y Madagascar.
Según Tomàs Marquès-Bonet, "los humanos somos primates. El estudio de cientos de genomas de primates no humanos, dada su posición filogenética, es muy valioso para los estudios evolutivos humanos, para entender mejor el genoma humano y las bases de nuestra singularidad, incluidas las bases de las enfermedades humanas, y para su futura conservación".
Estos estudios también han indicado que la genética de los primates no siempre se corresponde con su taxonomía. Encontramos varios casos en los que las relaciones entre especies de primates se describen más como redes complejas que como simples árboles ramificados.
Otro estudio profundiza en la evolución de las especies de babuinos, un grupo amplio y diverso de monos, mostrando que ha habido varios episodios de hibridación y flujo genético entre especies que no se habían reconocido anteriormente. Además, descubrimos que los babuinos amarillos del oeste de Tanzania son los primeros primates no humanos que han recibido aportaciones genéticas de tres linajes distintos. "Estos resultados sugieren que la estructura genética de la población y la historia de la introgresión entre linajes de babuinos es más compleja de lo que se pensaba, y eso demuestra que los babuinos son un buen modelo para la evolución de los humanos, los Neandertales y los Denisovanos". Afirma Jeffrey Rogers, profesor asociado del Centro de Secuenciación del Genoma Humano y del Departamento de Genética Molecular y Humana de la Facultad de Medicina de Baylor, que ha codirigido este estudio.
"Nuestros estudios proporcionan pistas sobre qué especies necesitan más urgentemente esfuerzos de conservación, y podrían ayudar a identificar las estrategias más eficaces para preservarlas", afirma Lukas Kuderna, primer autor de uno de los estudios.
Por último, el nuevo catálogo genómico ha reducido a la mitad el número de innovaciones genómicas que se creían exclusivamente humanas. Esta observación facilita la identificación de aquellas mutaciones no compartidas con los primates que, en consecuencia, pueden ser exclusivas de la evolución humana y de las características que nos hacen humanos.

Los investigadores del IBE y la UPF Lukas FK, Kuderna, Alejandro Valenzuela, David Juan, Arcadi Navarro y Esther Lizano también han participado en los estudios agrupados en esta edición especial de Science.
El otro consorcio del número especial, codirigido por Guojie Zhang, de la Universidad de Zhejiang, y Dong-dong Wu, del Instituto de Zoología de Kunming, con la colaboración de la IBE, ha llevado a cabo análisis genómicos comparativos de los genomas de referencia de 50 especies de primates, incluida la humana, y también ha contribuido con otros cuatro artículos a este número especial. Estos artículos se centran en las evoluciones genómicas y aportan nuevos conocimientos sobre el proceso de especiación de las especies de primates, las innovaciones genómicas relacionadas con la singularidad humana y la evolución social de los primates.
Artículos de referencia:
Kuderna, L.F.K et al., ‘A global catalog of whole-genome diversity from 233 primate species’ Science. May, 2023. DOI: 10.1126/science.abn7829
Sorensen, E, et al., ‘Genome-wide coancestry reveals details of ancient and recent male-driven reticulation in baboons’ Science. May 2023. DOI: 10.1126/science.abn8153
Gao, H, et al., ‘The landscape of tolerated genetic variation in humans and primates’ Science. May 2023. DOI: 10.1126/science.abn8197
